胆道闭锁(biliary atresia, BA)是新生儿及婴儿期阻塞性黄疸的主要病因之一,其以肝内、肝外胆道闭塞为主要特征,可导致不可控制、迅速发展的进行性肝纤维化,如不及时治疗,患儿常在1岁内死亡[1, 2]。BA的病因目前仍不清楚,可能与多种因素(如发育异常、遗传易感性、血运障碍、病毒感染、胆汁酸代谢异常、免疫及炎症反应等)有关[3, 4]。BA的发病率有明显的种族和地区差异,其中亚洲地区明显高于欧美地区[5, 6]。此外,有研究报道家庭中数名BA患儿,以及母亲与孩子同患BA的文献[7]。这提示BA的发病存在遗传因素的作用。Garcia-Barcelo等[8]于2010年对中国人群中BA患儿进行了首个全基因组关联分析(genome-wide association study, GWAS),发现了10个与BA相关性较大的单核苷酸多态性(single nucleotide polymorphism, SNP)位点,其中相关性最强的是rs17095355位点,分析显示该位点的OR值及其95%CI为1.77 (1.38~2.26),增加了BA的易患性。rs17095355位点位于ADD3基因上,胞嘧啶可转换为胸腺嘧啶(C>T),随后在泰国以及其他中国人群中的研究也发现该位点多态性显著增加了BA的易患风险,而Tsai等的研究结论与上述不同[9-14]。本研究拟对rs17095355位点多态性与胆道闭锁相关性研究进行Meta分析,为胆道闭锁的发病机制提供线索。
材料与方法 一、检索策略检索的数据库包括英文数据库(PubMed、EMbase、Web of Science)及中文数据库(中国知网、万方数据知识服务平台、维普期刊资源整合服务平台和中国生物医学文献服务系统)。检索时间截至2019年7月。英文检索词:“biliary atresia”OR“bile duct atresia”、“rs17095355”OR“ADD3”OR“XPNPEP1”、“polymorphism”OR“genetic variant”OR“genetic variation”等。中文检索词:“胆道闭锁”、“rs17095355或ADD3或XPNPEP1”、“多态性”等。
二、文献纳入及排除标准纳入标准:①人群研究或病例对照研究;②研究内容包括rs17095355位点与胆道闭锁相关性;③语种为中文或英文;④能够提供各个等位基因在病例组和对照组中的频数分布用于计算比值比(odds ratio, OR)。排除标准:①不能提供完整数据或数据存在明显错误的文献;②通信、综述、病例报道、会议摘要、期刊述评、专家意见;③未设立对照组。阅读标题和摘要,依据纳入及排除标准进行初步筛选,排除不符合要求的文献,通读入选文献后进行再次筛选。
三、信息提取和文献质量评价对纳入文献使用自拟提取表提取资料,提取信息包括文献特征(第一作者姓名、发表年份、研究人群等)以及病例组与对照组的例数、等位基因频率等。采用纽卡斯尔-渥太华(Newcastle-Ottawa scale, NOS)量表对纳入文献进行质量评价,内容包括:①研究人群选择:诊断是否明确(1分),病例是否具有代表性(1分),对照来源(1分),对照确定(1分);②组间可比性:设计和统计分析时考虑病例组和对照组的可比性(2分);③暴露因素测量:暴露因素确定(1分),暴露是否采用相同检测方法(1分),无应答率(1分)。总分9分,5分以上可视为高质量文献。整个过程由两位评价者独立完成,不一致处经讨论后决定。
四、统计学分析数据分析采用Stata 14.0软件。应用Q检验法(χ2检验)对所纳入的研究进行异质性检验,同时计算I2统计量,以P≤0.1为存在异质性。当同时满足P>0.1和I2≤40%时,采用固定效应模型,反之则采用随机效应模型[15]。OR值计算方法为实验组中某等位基因或基因型与其它等位基因或基因型之比除以对照组之比,OR=1提示该因素对疾病的发生不起作用,95%CI下限>1提示可能为危险因素,上限<1时提示可能为保护因素,上述结果均采用Stata 14.0计算。假定T等位基因为危险基因,分别计算在等位基因遗传模型(T vs.C)、显性基因遗传模型(TT+CT vs.CC)、隐性基因遗传模型(TT vs.CT+CC)下的合并OR值及其95%CI。并按样本量大小进行亚组分析。使用在线工具验证对照组基因频率分布是否符合H-W遗传平衡定律。并逐一剔除每一项研究进行敏感性分析。采用漏斗图观察发表偏倚,并用Egger's检验评价漏斗图的对称性,P>0.1时可判断漏斗图对称,无明显发表偏倚。
结 果 一、文献检索结果共检索到2019年7月以前发表的相关文献33篇,剔除重复文献后获得12篇,其中综述3篇,其它基因1篇,研究对象非人群1篇。排除上述不符合纳入标准的文献,最终纳入7篇文献,见图 1。

|
Download:
|
| 图 1 文献检索纳入排除流程图 Fig. 1 Flow diagram of study selecting | |
7篇文献发表时间为2010—2018年,共包括8组独立样本研究(Garcia-Barcelo等2010年的研究中包括两组独立样本),均为病例对照研究。7组研究对象为亚洲人群[8-13],1组研究对象为高加索人[14]。纳入文献中有2篇样本量≥1 000例[12, 14];3篇文献明确提及采用通用诊断标准[12-14],2篇文献中为已行Kasai手术患儿[10, 11];所有文献对照组基因分布均符合H-W遗传平衡定律;SNP检测方法包括Sanger直接测序法、Taqman探针法、基因芯片法;4篇文献对照组来源于健康人群[9-11, 14]。所纳入文献NOS评分均>5分。入选文献基本特征、质量评价及rs17095355位点等位基因分布情况见表 1。
|
|
表 1 入选文献基本特征和NOS评分及rs17095355位点等位基因分布情况 Table 1 Major characteristics and quality evaluations of eligible studies and the distribution of rs17095355 |
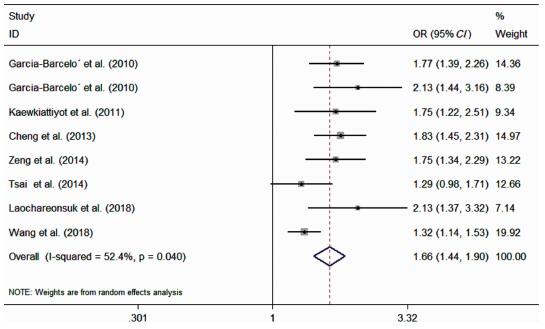
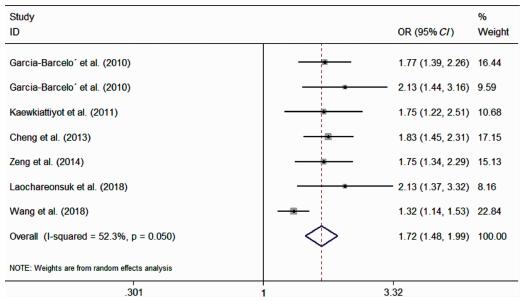
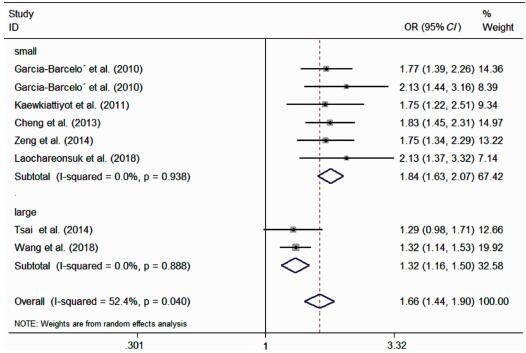
Meta分析结果显示,等位基因遗传模型下rs17095355位点多态性与胆道闭锁患病风险的合并OR值及95%CI为1.66(1.44~1.90),见图 2。亚洲人群中等位基因遗传模型下该位点多态性与胆道闭锁患病风险的合并OR值及95%CI为1.72(1.48~1.99),见图 3。对所有研究按样本量大小是否≥1 000人进行亚组分析后,异质性减少。在样本量<1 000人组和样本量≥1 000人组中,该位点均增加了BA的易患性,合并OR值及95%CI分别为1.84(1.63~2.07)和1.32(1.16~1.50),见图 4。其中4组研究提供了BA组与对照组具体的基因型分布例数[10-13],见表 2。在显性基因遗传模型(TT+CT vs. CC)、隐性基因遗传模型(TT vs. CT+CC)下rs17095355位点多态性均使胆道闭锁的患病风险增加,OR值及95%CI分别为1.66(1.38~2.01)和2.16(1.38~3.37),见表 3。
|
Download:
|
| 图 2 等位基因遗传模型下rs17095355位点多态性与BA关联的森林图 Fig. 2 Forest plots of rs17095355 polymorphism and BA in allele genetic model | |
|
Download:
|
| 图 3 亚洲人群中等位基因遗传模型下rs17095355位点多态性与BA关联的森林图 Fig. 3 Forest plots of rs17095355 polymorphism and BA in allele genetic model in Asian populations | |
|
Download:
|
| 图 4 按样本量大小进行亚组分析后等位基因遗传模型下rs17095355位点多态性与BA关联的森林图 Fig. 4 Forest plots of rs17095355 polymorphism and BA in allele genetic model by subgroup analysis | |
|
|
表 2 BA组与对照组基因型分布情况(n) Table 2 The distribution of genotypes in four studies(n) |
|
|
表 3 不同遗传模型下rs17095355位点与胆道闭锁的关联 Table 3 Meta-analysis of rs17095355 polymorphism and biliary atresia risk |
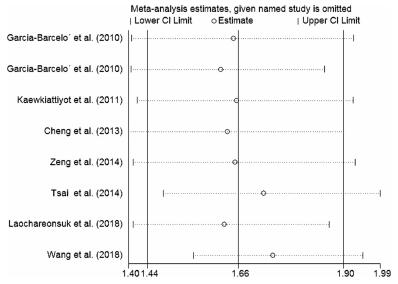
逐个去掉任何一项研究,对等位基因模型下rs17095355位点基因多态性与胆道闭锁关联性研究的Meta分析结果进行敏感性分析,发现对合并OR值的影响不大(图 5)。
|
Download:
|
| 图 5 敏感性分析 Fig. 5 Sensitivity analysis | |
对等位基因模型下rs17095355位点多态性与胆道闭锁关联性研究的Meta分析采用Egger's检验进行发表偏倚的统计学检验,发现存在发表偏倚(t=2.72,P=0.035)。
讨 论胆道闭锁的病因及发病机制至今尚未完全明确,包括遗传因素、血运障碍、病毒感染、胆汁酸代谢异常、免疫及炎症反应等多种因素[3, 4]。单核苷酸多态性是人类可遗传的变异中最常见的一种,占基因多态性的80%以上。基于对BA发病机制的探索和对遗传学认识的深入,以及基因分型技术的发展,越来越多的学者关注着基因、SNP与BA之间的关系,进行了诸多寻找和验证BA易感基因的研究[8-14]。本次Meta分析纳入了7篇关于rs17095355位点多态性与BA相关性的文献,共包括8组独立样本研究。其中1组研究对象为高加索人,剩余7组均为亚洲人,包括5组中国人群研究和2组泰国人群研究[8-14]。本Meta分析结果显示,在3种遗传模型包括等位基因遗传模型、显性基因遗传模型和隐性基因遗传模型下,rs17095355位点多态性均增加了胆道闭锁的患病风险,OR值及95%CI分别为1.66(1.44~1.90)、1.66(1.38~2.01)和2.16(1.38~3.37)。
8组研究均采用病例对照研究探讨了等位基因遗传模型下rs17095355位点与BA的相关性。Garcia-Barcelo等[8]于2010年进行了首个基于中国BA患儿的GWAS研究,筛选出了10个与BA相关性最强的SNPs,其中就包括10q24.2区的rs17095355位点,分析显示该位点OR值及95%CI为1.77 (1.38~2.26),差异有统计学意义(P=4.75×10-6)。随后独立样本(124名BA患儿和90名健康儿童)的验证结果中同样发现rs17095355位点与BA相关性最强,OR值及95%CI为2.13(1.42~3.20),差异有统计学意义(P=6.94×10-9)。此后关于亚洲人群中该位点与BA易感性的文献包括2011年Kaewkiattiyot等[10]及2018年Laochareonsuk等[11]在泰国人群,以及2013年Cheng等[9]、2014年Zeng等[13]和2018年Wang等[12]在中国人群中的研究。其中Wang等[12]研究总人数超过1 000人,计算的OR值及95%CI为1.33(1.15~1.54),差异有统计学意义(P=1.60×10-4),相较其他研究稍低。本Meta分析计算显示亚洲人群中该位点在等位基因遗传模型下的合并OR值及95%CI为1.72(1.48~1.99),与大部分研究结果相近。考虑到纳入研究中仅1组针对高加索人,其余均为亚洲人群的研究,故未对种族进行亚组分析。唯一以高加索人为研究对象的数据来自Tsai等,该研究总样本量超过1 000人,包括171名BA患儿与1 630名对照,结果发现rs7099604为10q24.2区与BA相关性最强的SNP位点,OR值及95%CI为1.45(1.09~1.92),差异有统计学意义(P=2.46×10-3),而非rs17095355位点,OR值及95%CI为1.29(0.98~1.71),差异有统计学意义(P=2.65×10-2),不同于亚洲人群[14]。已知BA发病率存在种族差异[5, 6],亚洲国家可达欧美国家的两倍[16]。亚洲人群与高加索人群中rs17095355位点OR值的差异可能与此因素有关[14]。7篇文章中4篇提供了BA组与对照组的基因型分布情况,结果发现在显性基因遗传模型、隐性基因遗传模型下rs17095355位点多态性均使胆道闭锁的患病风险增加,但纳入研究数量较少,结果的可信度受到限制[10-13]。考虑到该类研究中由于某些基因较低或较高的最小等位基因频率(minimal allele frequency, MAF)值及样本量的不足等因素很容易出现统计结果的假阳性、假阴性,因此本研究以样本量大小是否超过1 000人对研究规模进行了亚组分析,结果发现两组中该位点均增加了BA的易感性,而异质性减少,提示样本量可能是异质性的来源。因此,更可靠的结果有待不同种族中更多样本量大、数据详细的研究来验证。
值得注意的是,目前对SNP的研究不仅集中在疾病易感性,随着认识的深入,越来越多的研究也开始关注SNP对基因功能的影响与疾病的关系[17, 18]。rs17095355位点位于10q24区的ADD3基因上,同样位于该区XPNPEP1基因的上游。ADD3基因编码一种膜骨架蛋白即内收蛋白3,可促进肝脏、胆道上皮细胞细胞间连接、黏附,调节细胞的迁移及黏附,而胆小管细胞的收缩需要肌动蛋白-肌球蛋白相互作用,若该机制障碍可出现胆汁淤积[19]。XPNPEP1表达于肝胆上皮细胞中,编码氨肽酶P1,该酶特异性地催化N端与倒数第二位脯氨酸相连氨基酸的裂解,参与胆汁酸的代谢、分泌及炎症过程[10]。免疫组化研究显示,XPNPEP1在胆道上皮细胞及肝细胞中都有表达,而ADD3特异性地表达于胆道上皮细胞中,在肝细胞中很少或不表达[14]。此外,有研究者发现,XPNPEP1基因敲除的斑马鱼很少或不发生胆道异常,但在ADD3敲除后可存在肝内胆道结构的缺陷[14]。关于rs17095355位点多态性是否影响ADD3或XPNPEP1基因的功能,数名研究者进行了探讨[8-10, 14]。Garcia-Barcelo等[8]通过生物信息学分析(Genomatix suite)发现,rs17095355位点的C>T改变了转录因子结合位点(transcription factor binding sites, TFBSs),然而这一转换对包含该位点及其周围的序列并无功能学影响。Tsai等[14]采用qPCR法检测正常对照、肝内胆汁淤积和BA肝脏中XPNPEP1和ADD3的表达,三组患者XPNPEP1表达无差异,而BA与正常对照组之间的ADD3表达水平有差异(P < 0.001)。Cheng等[9]通过生物信息学和体内基因表达研究发现,与BA发病相关的调控性SNPs与ADD3基因在肝脏中的蛋白表达有关系,而XPNPEP1则没有。这些均提示该位点可能通过影响ADD3的功能表达在BA发病中发挥作用。大多数研究并未涉及rs17095355位点与BA患儿临床及病理特征是否存在关系。仅Kaewkiattiyot等[10]将BA患儿分为无黄疸组(TB < 2 mg/dL)和持续黄疸组(TB≥2 mg/dL),结果发现两组间基因型及rs17095355位点表达并无统计学差别。rs17095355多态性是否以及如何影响ADD3表达,是否与BA患儿临床特征及病理特点存在关系,还需要进一步探索。
本研究存在一定局限性。①胆道闭锁的发病机制复杂,可能存在基因-基因、基因-环境的交互作用。研究者们还对rs2501577、rs10509906等SNP位点与胆道闭锁患病风险的相关性进行了分析,并发现了一些具有意义的单体型,鉴于其它位点的意义及相关研究数量有限,本研究只针对rs17095355位点进行了分析。此外,有学者认为胆道闭锁是在遗传学背景的基础上,在环境因素作用下诱发的异常免疫反应[20, 21]。有报道异卵或同卵双胞胎中只有1人患BA的情况,但目前关于胆道闭锁的基因多态性研究中普遍缺乏病毒感染等环境因素资料。②本研究检索了几个主要的中英文数据库,未包括其他语言撰写的文献,使纳入研究的全面性受到了一定影响。日本胆道闭锁发病率相对较高,但所检索文献除中国、泰国外,无亚洲其他国家的相关研究数据,不能揭示亚洲其他国家rs17095355位点基因多态性与胆道闭锁的关系。本研究的相关结果尚需在亚洲其他国家,以及美洲、欧洲等开展严格设计的病例对照或前瞻性研究加以验证。③一般认为BA可分为两大类型:胚胎型(先天型)和围生期型(获得型),两者发病机制有所不同。纳入文献中约50%作者未注明胆道闭锁类型[10-12, 14]。胆道闭锁女孩多于男孩,但纳入文献无充足数据对性别进行亚组分析。4篇文献提供了数据用于在显性模型及隐性模型下进行比较,但样本数量较少。④Cochrane手册中认为在纳入研究数量少于10个时,Egger检验的效能和敏感性都受到限制,本研究在全面检索后纳入8组研究,此时检验效能受到限制,因此尽管Egger检验提示发表偏倚,本研究结果仍有参考价值。
总之,Meta分析发现rs17095355位点多态性与胆道闭锁患病风险相关,T等位基因可能是发生胆道闭锁的危险因素,特别是在亚洲人群中。然而以上结果仍需要来自亚洲其他地区以及美洲、欧洲等不同种族的更大规模的研究来将现有的数据库建立得更加的庞大、丰富。此外,rs17095355位点是否以及如何影响相关基因的表达及功能,是否与BA患儿临床及病理特征存在关联,还需要进一步研究。
| 1 |
Kilgore A, Mack CL. Update on investigations pertaining to the pathogenesis of biliary atresia[J]. Pediatr Surg Int, 2017, 33(12): 1233-1241. DOI:10.1007/s00383-017-4172-6. |
| 2 |
Bezerra JA, Wells RG, Mack CL, et al. Biliary atresia:clinical and research challenges for the twenty-first century[J]. Hepatology, 2018, 38(3): 1163-1173. DOI:10.1002/hep.29905. |
| 3 |
詹江华, 余晨. 胆道闭锁发病机制研究概述与启示[J]. 临床小儿外科杂志, 2018, 17(11): 801-804. DOI:10.3969/j.issn.1671-6353.2018.11.001. J H, Yu C. An overview and enlightenment of the pathogenesis of biliary atresia[J]. J Clin Ped Sur, 2018, 17(11): 801-804. DOI:10.3969/j.issn.1671-6353.2018.11.001. |
| 4 |
Wehrman A, Waisbourd-Zinman O, Wells RG, et al. Recent advances in understanding biliary atresia[J]. F1000Res, 2019, 8. DOI:10.12688/f1000research.16732.1. |
| 5 |
Jimenez-Rivera C, Jolin-Dahel KS, Fortinsky KJ, et al. International incidence and outcomes of biliary atresia[J]. J Pediatr Gastroenterol Nutr, 2013, 56(4): 344-354. DOI:10.1097/MPG.0b013e318282a913. |
| 6 |
Lee KJ, Kim JW, Moon JS, et al. Epidemiology of biliary atresia in Korea[J]. J Korean Med Sci, 2017, 32(4): 656-660. DOI:10.3346/jkms.2017.32.4.656. |
| 7 |
Cameron-Christie SR, Wilde J, Gray A, et al. Genetic investigation into an increased susceptibility to biliary atresia in an extended New Zealand Maori family[J]. BMC Med Genomics, 2018, 11(1): 121. DOI:10.1186/s12920-018-0440-0. |
| 8 |
Garcia-Barcelo MM, Yeung MY, Miao XP, et al. Genome-wide association study identifies a susceptibility locus for biliary atresia on 10q24[J]. Human Molecular Genetics, 2010, 19(14): 2917-2925. DOI:10.1093/hmg/ddq196. |
| 9 |
Cheng G, Tang CS, Wong EH, et al. Common genetic variants regulating ADD3 gene expression alter biliary atresia risk[J]. J Hepatol, 2013, 59(6): 1285-1291. DOI:10.1016/j.jhep.2013.07.021. |
| 10 |
Kaewkiattiyot S, Honsawek S, Vejchapipat P, et al. Association of X-prolyl aminopeptidase 1 rs17095355 polymorphism with biliary atresia in Thai children[J]. Hepatol Res, 2011, 41(12): 1249-1252. DOI:10.1111/j.1872-034X.2011.00870.x. |
| 11 |
Laochareonsuk W, Chiengkriwate P, Sangkhathat S. Single nucleotide polymorphisms within Adducin 3 and Adducin 3 antisense RNA1 genes are associated with biliary atresia in Thai infants[J]. Pediatr Surg Int, 2018, 34(5): 515-520. DOI:10.1007/s00383-018-4243-3. |
| 12 |
Wang Z, Xie X, Zhao J, et al. The intragenic epistatic association of ADD3 with biliary atresia in Southern Han Chinese population[J]. Biosci Rep, 2018, 38(3): BSR20171688. DOI:10.1042/BSR20171688. |
| 13 |
Zeng S, Sun P, Chen Z, et al. Association between single nucleotide polymorphisms in the ADD3 gene and susceptibility to biliary atresia[J]. PLoS One, 2014, 9(10): e107977. DOI:10.1371/journal.pone.0107977. |
| 14 |
Tsai EA, Grochowski CM, Loomes KM, et al. Replication of a GWAS signal in a Caucasian population implicates ADD3 in susceptibility to biliary atresia[J]. Hum Genet, 2014, 133(2): 235-243. DOI:10.1007/s00439-013-1368-2. |
| 15 |
Magosi LE, Goel A, Hopewell JC, et al. Identifying systematic heterogeneity patterns in genetic association meta-analysis studies[J]. PLoS Genetics, 2017, 13(5): e1006755. DOI:10.1371/journal.pgen.1006755. |
| 16 |
Zhan J, Feng J, Chen Y, et al. Incidence of biliary atresia associated congenital malformations:A retrospective multicenter study in China[J]. Asian J Surg, 2017, 40(6): 429-433. DOI:10.1016/j.asjsur.2016.04.003. |
| 17 |
Shan Y, Shen N, Han L, et al. MicroRNA-499 Rs3746444 polymorphism and biliary atresia[J]. Dig Liver Dis, 2016, 48(4): 423-428. DOI:10.1016/j.dld.2015.11.014. |
| 18 |
Sheu MJ, Hsieh MJ, Chiang WL, et al. Fibroblast growth factor receptor 4 polymorphism is associated with liver cirrhosis in hepatocarcinoma[J]. PLoS One, 2015, 10(4): e122961. DOI:10.1371/journal.pone.0122961. |
| 19 |
Tang V, Cofer ZC, Cui S, et al. Loss of a candidate biliary atresia susceptibility gene, add3a, causes biliary developmental defects in zebrafish[J]. J Pediatr Gastroenterol Nutr, 2016, 63(5): 524-530. DOI:10.1097/MPG.0000000000001375. |
| 20 |
Averbukh LD, Wu GY. Evidence for viral induction of biliary atresia:a review[J]. J Clin Transl Hepatol, 2018, 6(4): 410-419. DOI:10.14218/JCTH.2018.00046. |
| 21 |
Asai A, Miethke A, Bezerra JA. Pathogenesis of biliary atresia:defining biology to understand clinical phenotypes[J]. Nat Rev Gastroenterol Hepatol, 2015, 12(6): 342-352. DOI:10.1038/nrgastro.2015.74. |
 2020, Vol. 19
2020, Vol. 19


