2. 陕西省人民医院新生儿科(陕西省西安市, 710068)
2. Department of Neonatology, Shaanxi Provincial People's Hospital, Xi'an 710068, China
肝母细胞瘤(hepatoblastoma, HB)是儿童最常见的肝脏恶性肿瘤,约占儿童肝脏原发性恶性肿瘤的80%[1]。随着新生儿重症监护技术的不断进步,极低出生体重儿存活率得到了显著提高,而肝母细胞瘤的年发病率也呈上升趋势[2]。近年来,手术切除+新辅助化疗的联合治疗模式极大地提高了肝母细胞瘤患儿的长期生存率(5年生存率已达80%以上),但肝母细胞瘤晚期或化疗耐药的患儿预后仍然很差[3]。虽然铂类药物治疗肝母细胞瘤的疗效显著,但容易造成长期不良反应(如耳毒性和肾毒性),给肝母细胞瘤患儿家庭造成沉重的经济和心理负担[1, 4]。迄今,肝母细胞瘤的病因及发病机制仍不明确,寻找肝母细胞瘤早期的发病机制及潜在治疗靶点对于肝母细胞瘤患儿早期诊疗及预后改善至关重要。
2017年发表在Hepatology杂志上的一项研究显示,SALL4基因在肝母细胞瘤组织中的表达水平较癌旁正常肝组织呈显著升高趋势,非编码RNA中let-7家族的表达水平则呈显著下降趋势,其中以let-7b最为显著。此外该研究还发现SALL4基因的高表达及let-7b的低表达与肝母细胞瘤的不良预后及高发病风险密切相关[4]。尽管如此,国内外针对let-7b在肝母细胞瘤中的可能作用机制的研究仍比较罕见,因此本研究通过体外实验,初步分析let-7b在肝母细胞瘤中的可能作用及作用机制。
材料与方法 一、患者标本及相关病理资料的收集从西安交通大学第二附属医院病理科收集31对肝母细胞瘤及对应的瘤旁正常肝组织蜡块,标本制作时间从2010年1月至2018年1月,均为接受手术切除治疗后经10%中性福尔马林固定得到的石蜡标本,并经过病理学检查证实。于本院病案室查阅入选患者相关临床病理学资料,具体包括:性别、年龄、肿瘤直径大小、血清甲胎蛋白(alpha fetoprotein, AFP)水平、病理类型和PRETEXT分期等。
二、细胞培养HL-7702正常肝细胞与肝母细胞瘤细胞株(HepG2和HuH6)的培养环境为含10%FBS及1%双抗的DMEM高糖培养基,37℃,5%CO2,每2~3 d换液1次。let-7b抑制剂由上海吉玛制药技术有限公司合成。
三、PCR检测let-7b的表达水平根据石蜡RNA提取试剂盒(美国omega公司)说明书中操作步骤提取总RNA。逆转录let-7b,引物为:5′-GTCGTATCCAGTGCGTGTCGTGGAGTCGGCAATTGCACTGGATACGACAACCACA-3′;逆转录反应体系:5×primer Script Buffer 2μL、RT Enzyme Mix 0.5μL、let-7b逆转录引物0.5μL、RNA(200ng/μL)1μL、RNAase free ddH2O 6μL。反应条件:42℃ 15 min×3 cycles;85℃ 5sec。逆转录得到cDNA,并测定其浓度,然后稀释至50 ng/μL,-20℃冻存。Real-time PCR反应部分,let-7b引物为:F:5′-ATCCAGTGCGTGTCGTG-3′;R:5′-TGCTTGAGGTAGTAG-GTTG-3′。Real-time PCR反应体系:SYBR premix ExTaqTM Ⅱ(2×)10 μL、F-let-7b-primer 1 μL、R-let-7b-primer 1 μL、cDNA(100 ng/μL)1 μL、ddH2O 7 μL。反应条件:第一步,95℃30 s;第二步,95℃5 s,60℃30 s,40cycles循环完成后绘制溶解曲线。
四、MTT法检测细胞增殖转染let-7b/anti-let-7b和miR-ctrl/miR-CN的HepG2细胞,在转染后24 h、48 h及72 h,于避光条件下每孔加入20 μL浓度为5 mg/mL的MTT溶液。4 h后弃掉旧培养基,每孔加入DMSO 150 μL,室温下缓慢震荡约10 min,用酶标仪于490 nm处检测各孔的OD值,并记录结果。
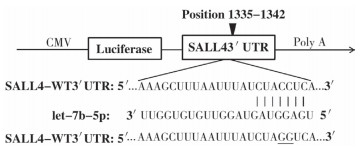
五、双荧光素酶报告基因实验通过TargetScan(http://targetscan.org/)预测到let-7b和SALL4具有良好的靶向关系。对SALL4的3′-UTRs种子区突变两个碱基位点,使之成为突变型序列(图 1)。
|
Download:
|
| 图 1 SALL4野生型和突变型碱基序列 Fig. 1 SALL4 wild/mutant type base sequences | |
实验分为四组:无处理细胞组、let-7b与空载体共转染组、let-7b与SALL4-WT共转染组、以及let-7b与SALL4-MT共转染组;将HEK 293细胞接种于96孔板中,采用脂质体(lipofectamine)2000试剂盒进行转染。
双荧光素酶报告基因的检测:待培养24 h后,每孔更换新鲜的完全培养基75 μL。每孔加入75 μL Dual-Glo Luciferase Buffer与Dual-Glo Luciferase Reagent的混合液,室温下静置10 min后于化学发光仪中检测荧光。每孔加入75 μL Dual-Glo stop Buffer与Dual-Glo stop Reagent的混合液,室温下静置10 min后于化学发光仪中检测荧光。
六、统计学处理所有数据均采用SPSS17.0进行统计学分析,由于本研究中的计量资料基本服从或近似服从正态分布,故采用(x±s)进行统计描述,两组样本间的比较采用独立样本t检验。采用χ2检验或Fisher精确概率法分析let-7b与肝母细胞瘤临床病理常数的关联性;采用Spearman秩相关分析let-7b与SALL4表达水平之间的相关性。采用Kaplan-Meier生存曲线分析不同let-7b与SALL4表达水平组肝母细胞瘤患者总体生存率的差异。P < 0.05为差异具有统计学意义。
结 果 一、纳入研究的肝母细胞瘤患儿基本情况纳入本研究的31例肝母细胞瘤患儿中,男20例,女11例,年龄1个月至12岁,平均年龄(1.46±0.72)岁。PRETEXT分期:Ⅰ期3例、Ⅱ期9例、Ⅲ期13例、Ⅳ期6例。23例(74.2%)以腹部包块就诊,临床表现4例(12.9%)为纳差、腹胀,2例(6.5%)为腹痛,2例(6.6%)为黄疸、消瘦,其余无特殊症状。病理类型:上皮型24例(其中胎儿型12例、胚胎型10例、粗大小梁型1例、混合上皮型1例),混合型7例。低危组8例,中危组12例,高危组11例。
let-7b表达水平与肝母细胞瘤患者的年龄、性别、病理类型、AFP、危险度等病理参数之间无显著关联性,但是let-7b在肝母细胞瘤组织中的表达水平与PRETEXT分期存在统计学关联(P < 0.05)。见表 1。
|
|
表 1 let-7b与肝母细胞瘤患者临床基本特征的关联性分析 Table 1 Correlation analysis of let-7b and basic clinical characteristics of children with hepatoblastoma |
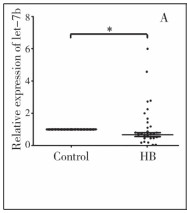
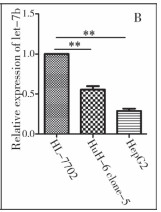
采用qRT-PCR检测let-7b在正常肝细胞(HL-7702)、HB细胞系(HuH-6 clone-5和HepG2)及31例HB组织标本中的表达情况,结果显示let-7b在HB细胞系及HB组织中的表达显著下调(图 2A,2B)。let-7b在2种肝母细胞瘤细胞系中,在HepG2中的表达水平最低,因此选择HepG2进行后续的功能研究实验。
|
Download:
|
| 图 2A let-7b在HB组织中的表达下调 Fig. 2A Expression level of let-7b in HB tissue | |
|
Download:
|
| 图 2B let-7b在HB细胞系中的表达降低 Fig. 2B Expression level of let-7b in HB cell lines | |
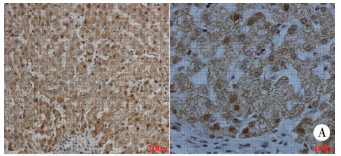
采用免疫组化法检测SALL4在肝母细胞瘤组织及对应的瘤旁正常肝组织中的表达水平,SALL4在肝母细胞瘤组织中的表达水平较瘤旁正常肝组织明显升高。图 3A可见SALL4蛋白主要表达于瘤细胞胞浆内,呈棕褐色颗粒状,以弥漫性分布为主,图 3B可见SALL4蛋白在正常肝组织中基本不表达。
|
Download:
|
| 图 3A SALL4在HB组织中的表达 Fig. 3A Expression of SALL4 protein in hepatoblastoma tissue | |

|
Download:
|
| 图 3B SALL4在瘤旁正常肝组织中的表达 Fig. 3B Expression of SALL4 protein in adjacent non-tumor tissues | |
与对照组相比,上调let-7b的HepG2细胞增殖活性在48 h和72 h时均明显受到抑制。抑制内源性let-7b的表达后,与miR-ctrl组相比,HepG2细胞的活性在48 h和72 h时有明显升高(P < 0.05),提示let-7b能够抑制HepG2细胞的增殖活性。
五、let-7b对HepG2细胞凋亡的影响采用流式细胞仪检测各组转染后HepG2细胞凋亡情况。let-7b过表达组和miR-ctrl组相比,let-7b组早期凋亡率显著高于对照组(P < 0.01)。抑制剂anti-miR let-7b组和anti-NC组相比,早期凋亡率无明显差异(P>0.05)。
六、let-7b与HepG2细胞周期S期/G2期转变的关系采用流式细胞仪检测转染后各组HepG2细胞系所处的周期情况。对HepG2细胞分别转染let-7b和空质粒载体,24 h后采用流式细胞仪检测两组细胞周期改变情况,结果显示转染let-7b的HepG2细胞S期/G2期受到显著阻滞(P=0.016)。将let-7b抑制剂转染至HepG2细胞,可见其周期S期的细胞比例较对照组有所下降,而G2期/M期的细胞比例升高(P>0.05)。
七、let-7b对SALL4野生型和突变型报告基因荧光活性的影响如图 4所示,经过共转染let-7b和SALL4-WT重组质粒的HEK293细胞荧光活性显著降低(P=0.015),而共转染let-7b和SALL4-MT组的HEK293细胞荧光活性无显著变化(P>0.05), 提示let-7b能够直接结合SALL4的3′-UTRs区域,发挥潜在的靶向调控作用。
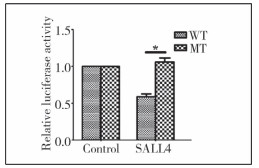
|
Download:
|
| 图 4 HEK293细胞中的荧光活性检测结果 Fig. 4 Activity of luciferase assay in HEK293 cells | |
采用半定量积分法,每张切片随机选取5个视野,高倍镜下计数500个细胞,参照Axiotis等[5]制定的标准,由2名病理医生采用双盲法分别对同一切片的染色进行评分,对结果进行复核。31例肝母细胞瘤患者中let-7b在瘤组织中的表达下调20例;SALL4蛋白在肝母细胞瘤组织中表达强阳性15例,阳性8例、弱阳性7例,阴性1例。let-7b在HB组织标本中与SALL4的表达存在统计学负相关(r=-0.716,P < 0.001),随着let-7b的表达水平降低,SALL4的蛋白表达水平呈现逐渐升高趋势。
九、let-7b对SALL4蛋白表达水平的影响在肝母细胞瘤细胞系HepG2中上调let-7b的表达水平后,SALL4蛋白的表达水平呈明显下降趋势,而抑制内源性的let-7b后,SALL4蛋白的表达水平较对照组呈上升趋势,提示在HepG2细胞中let-7b对SALL4蛋白的表达水平有显著抑制作用(图 5)。

|
Download:
|
| 图 5 SALL4蛋白在HepG2细胞中的表达水平 Fig. 5 Expression level of SALL4 protein in HepG2 cells | |
如图 6A所示,let-7b低表达组HB患儿的总体生存率显著低于let-7b高表达组(P=0.017)。如图 6B所示,SALL4阳性组HB患者的总体生存率显著低于SALL4阴性和弱阳性组(P=0.034)。
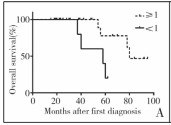
|
Download:
|
| 图 6A let-7b低表达与高表达组HB患者的生存曲线 Fig. 6A Survival curves of low versus high let-7b expression in HB patients | |
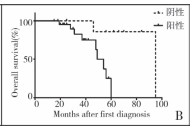
|
Download:
|
| 图 6B SALL4低表达与高表达组HB患者的生存曲线 Fig. 6B Survival curves of low versus high SALL4 expression in HB patients | |
本研究结果显示在肝母细胞瘤组织中let-7b的表达水平较瘤旁组织呈显著下降趋势,而SALL4的蛋白水平呈显著上调趋势,与Sumazin等[4]筛查得到的结果一致。我们检测了let-7b在肝母细胞瘤细胞(HepG2和Huh6)和正常肝细胞(HL-7702)中的表达水平差异,也发现let-7b在肝母细胞瘤细胞中的表达水平显著低于正常肝细胞,且let-7b在HepG2中的表达水平最低,因此我们选择HepG2细胞进行后续的功能及机制验证探索实验。本研究首次发现let-7b在肝母细胞瘤组织中的表达水平与PRETEXT分期存在统计学相关性,let-7b低表达组肝母细胞瘤患者的总体生存率显著低于let-7b高表达组。Sumazin等[4]发现let-7b的低表达与肝母细胞瘤的不良预后密切相关。本研究发现PRETEXTⅢ/Ⅳ期肝母细胞瘤患者中let-7b降低的比例(17/19)较PRETEXTⅠ/Ⅱ期患者(4/12)更高。而PRETEXTⅢ/Ⅳ期肝母细胞瘤患者较PRETEXTⅠ/Ⅱ期患者预后不佳这一结论已经基本明确,这也能从侧面证明let-7b的低表达与肝母细胞瘤的不良预后密切相关[4]。但纳入本研究的肝母细胞瘤患者总样本数仅31例,故后续仍需要进一步扩大临床样本量来验证。
研究发现let-7家族(包括let-7b)在肝癌等恶性肿瘤的发生及发展过程中发挥着抑癌基因样作用[6-8],且let-7b表达下调与前列腺癌、肝母细胞瘤的不良预后密切相关[4, 9]。let-7过表达可抑制肝癌干细胞增殖、体外成球及体内成瘤能力,而下调let-7的表达水平有促进肝癌干细胞增殖的作用[10]。上调let-7b的表达水平还可抑制胶质瘤干细胞样细胞的自我更新能力和干细胞特性的维持[11]。本研究结果表明上调let-7b的表达水平,可通过促进HepG2细胞早期凋亡,进而显著抑制HepG2细胞的增殖活性。抑制内源性let-7b的表达,可促进细胞的增殖,抑制细胞的凋亡。
为进一步研究let-7b抑制HepG2细胞增殖活性的机制,我们利用生物学信息软件预测得知SALL4与let-7b存在潜在的靶向关系。2013年发表在新英格兰医学杂志上的一项研究发现:SALL4高表达的肝细胞癌具有更强的侵袭性,通过敲除SALL4或阻断SALL4的表达水平,可促进肝癌细胞凋亡及抑制其肿瘤形成[12]。SALL4可作为胚胎干细胞的特异性标记物,参与调节肿瘤细胞的恶性生物学表型[13]。研究发现SALL4在肝母细胞瘤组织中表达水平异常增高,且与肝母细胞瘤的不良预后密切相关[4, 14]。本研究中通过免疫组化法,检查SALL4蛋白在肝母细胞瘤组织中的表达水平,发现与瘤旁组织相比,SALL4蛋白在肝母细胞瘤组织中的表达水平呈显著上调趋势,SALL4阳性组肝母细胞瘤患者的总体生存率显著低于SALL4阴性和弱阳性组(P=0.034),与上述研究结果一致。本研究结果还提示肝母细胞瘤组织中let-7b的表达水平与SALL4的表达水平呈显著负相关,双萤光素酶报告基因实验结果也证实let-7b与SALL4有良好的靶向关系。我们上调HepG2细胞中let-7b的表达水平后,SALL4蛋白的表达水平可被显著下调,而抑制let-7b的表达水平后则可以逆转这个现象。本研究结果提示在肝母细胞瘤中let-7b可能通过靶向调控SALL4的表达水平影响其恶性生物学行为。SALL4与转录因子包括SOX2、Oct3/4、Nanog等,这些因子相互作用共同维系了肿瘤干细胞(cancer stem cell, CSC)的多功能性,是维持CSC无限增殖、自我更新、多向分化的重要调控基因[15]。Bmi-1和PTEN已被证实是SALL4下游的直接靶基因[16, 17]。而SALL4具体通过下游的哪条通路在肝母细胞瘤中发挥作用尚需要我们进一步研究。
现有的研究表明肝母细胞瘤可表现出不同的组织学表型和临床行为,可能是由不同分化程度的肝干细胞或早期肝前体细胞增殖产生[4]。肝母细胞瘤中也被证实存在肿瘤干细胞样细胞(也称侧群细胞,side polulation cells, SP)的存在,接种到小鼠体内后与主群细胞(main polulation cells, MP)相比有更强的成瘤能力[18]。let-7b和SALL4均为干细胞标记物,在肝母细胞瘤中let-7b是否可以通过靶向调控SALL4影响肝母细胞瘤干细胞生物学特性,进而影响肝母细胞瘤的恶性表型还需要进一步研究。
| 1 |
Spector LG, Birch J. The epidemiology of hepatoblastoma[J]. Pediatr Blood Cancer, 2012, 59(5): 776-779. DOI:10.1002/pbc.24215. |
| 2 |
Bissig-Choisat B, Kettlun-Leyton C, Legras XD, et al. Novel patient-derived xenograft and cell line models for therapeutic testing of pediatric liver cancer[J]. J Hepatol, 2016, 65(2): 325-333. DOI:10.1016/j.jhep.2016.04.009. |
| 3 |
Hooks KB, Audoux J, Fazli H, et al. New insights into diagnosis and therapeutic options for proliferative hepatoblastoma[J]. Hepatology, 2018, 68(1): 89-102. DOI:10.1002/hep.29672. |
| 4 |
Sumazin P, Chen Y, Treviño LR, et al. Genomic analysis of hepatoblastoma identifies distinct molecular and prognostic subgroups[J]. Hepatology, 2017, 65(1): 104-121. DOI:10.1002/hep.28888. |
| 5 |
Axiotis CA, Monteagudo C, Merino MJ, et al. Immunohistochemical detection of P-glycoprotein in endometrial adenocarcinoma[J]. Am J Pathol, 1991, 138(4): 799-806. |
| 6 |
Wang Z, Xu L, Hu Y, et al. miRNA let-7b modulates macrophage polarization and enhances tumor-associated macrophages to promote angiogenesis and mobility in prostate cancer[J]. Sci Rep, 2016, 6: 25602. DOI:10.1038/srep25602. |
| 7 |
Tian Y, Hao S, Ye M, et al. MicroRNAs let-7b/i suppress human glioma cell invasion and migration by targeting IKBKE directly[J]. Biochem Biophys Res Commun, 2015, 458(2): 307-312. DOI:10.1016/j.bbrc.2015.01.105. |
| 8 |
Han X, Chen Y, Yao N, et al. MicroRNA let-7b suppresses human gastric cancer malignancy by targeting ING1[J]. Cancer Gene Ther, 2015, 22(3): 122-129. DOI:10.1038/cgt.2014.75. |
| 9 |
Saffari M, Ghaderian SMH, Omrani MD, et al. The Association of miR-let 7b and miR-548 with PTEN in Prostate Cancer[J]. Urol J, 2019, 16(3): 267-273. DOI:10.22037/uj.v0i0.4564. |
| 10 |
Jin B, Wang W, Meng XX, et al. Let-7 inhibits self-renewal of hepatocellular cancer stem-like cells through regulating the epithelial-mesenchymal transition and the Wnt signaling pathway[J]. BMC Cancer, 2016, 16(1): 863. DOI:10.1186/s12885-016-2904-y. |
| 11 |
Tian Y, Hao S, Ye M, et al. MicroRNAs let-7b/i suppress human glioma cell invasion and migration by targeting IKBKE directly[J]. Biochem Biophys Res Commun, 2015, 458(2): 307-312. DOI:10.1016/j.bbrc.2015.01.105. |
| 12 |
Yong KJ, Gao C, Lim JS, et al. Oncofetal gene SALL4 in aggressive hepatocellular carcinoma[J]. N Engl J Med, 2013, 368(24): 2266-2276. DOI:10.1056/NEJMc1308785. |
| 13 |
Tatetsu H, Kong NR, Chong G, et al. SALL4, the missing link between stem cells, development and cancer[J]. Gene, 2016, 584(2): 111-119. DOI:10.1016/j.gene.2016.02.019. |
| 14 |
Zhou S, Venkatramani R, Gomulia E, et al. The diagnostic and prognostic value of SALL4 in hepatoblastoma[J]. Histopathology, 2016, 69(5): 822-830. DOI:10.1111/his.13005. |
| 15 |
Tanimura N, Saito M, Ebisuya M, et al. Stemness-related factor Sall4 interacts with transcription factors Oct-3/4 and Sox2 and occupies Oct-Sox elements in mouse embryonic stem cells[J]. J Biol Chem, 2013, 288(7): 5027-5038. DOI:10.1074/jbc.M112.411173. |
| 16 |
Yang J, Chai L, Liu F, et al. Bmi-1 is a target gene for SALL4 in hematopoietic and leukemic cells[J]. Proc Natl Acad Sci U S A, 2007, 104(25): 10494-10499. DOI:10.1073/pnas.0704001104. |
| 17 |
Liu C, Wu H, Li Y, et al. SALL4 suppresses PTEN expression to promote glioma cell proliferation via PI3K/AKT signaling pathway[J]. J Neurooncol, 2017, 135(2): 263-272. DOI:10.1007/s11060-017-2589-3. |
| 18 |
Hayashi S, Fujita K, Matsumoto S, et al. Isolation and identif ication of cancer stem cells from a side populat ion of a human hepatoblastoma cell line, HuH-6 clone-5[J]. Pediatr Surg Int, 2011, 27(1): 9-16. DOI:10.1007/s00383-010-2719-x. |
 2020, Vol. 19
2020, Vol. 19


